VMD基础——atomselect命令
你可以用VMD的atomselect命令来编辑原子属性。下面的例子会告诉你怎样操作。
1 当确定晶体分子是顶层的分子后(如果不是,双击T)。 用Graphics —— Representations.菜单打开Graphical Representations窗口。
PDB B-因子范围。PDB 文件中“B”的范围通常储存晶体结构的“温度因子”,在 VMD 中读入“Beta”范围。由于我们目前的重点不在此,我们可以利用这个范围来存储我们自己的数字值。VMD 有一个“Beta”上色方法,它可以根据原子的 B 因子给它们上色。当用不同的原子来替换 B 值,你就可以控制不同原子的颜色了。当你想要展示通过计算得出来的系统属性时,这种方法就很有用了。但是这种方法在VMD 之外是不能应用的。
2 在窗口顶部的Selected Molecule下拉菜单中选择“crystal”的分子, 在atom selection中键入protein。把Coloring Method 设为 Beta,把Drawing Method设为VDW.。分子大部分会显示出红色和绿色小球的集合。
现在你将学到VMD中一个非常重要的Tcl命令:
atomselect molid selection – creates a new atom selection
对于atomselect,首先要讲的是molecule ID(主窗口的最左边),其次是文本原子选择,就PDB B-因子范围。PDB 文件中“B”的范围通常储存晶体结构的“温度因子”,在 VMD 中读入“Beta”范围。由于我们目前的重点不在此,我们可以利用这个范围来存储我们自己的数字值。VMD 有一个“Beta”上色方法,它可以根据原子的 B 因子给它们上色。当用不同的原子来替换 B 值,你就可以控制不同原子的颜色了。
当你想要展示通过计算得出来的系统属性时,这种方法就很有用了。但是这种方法在VMD 之外是不能应用的。像你在第一单元所学的用来描述图形显示方法的文本原子选择。你要学习的Atomselect,返回的选择是一个命令。
3 在Tk控制台窗口键入crystal [atomselect top "all"], 这个选择包括了分子中所有原子,并把所选赋予变量crystal。我们用“top”来指代顶层分子,不用molecule ID(是个数字,不好记)。Atomselect得出的结果是一个功能。因此,$crystal是一个针对“all”所指代内容执行的功能。
4 键入$crystal num. 把num赋给所选一个原子, 返回的是原子在这个选择中的序号 。检查一下这个序号是不是与分子中原子的序号相同(可以从main窗口中读取)。
5 键入$crystal set beta 0.。 这样就把所有原子的“beta”域设为0。当你这样做的时候,你会观察到屏幕上原子的颜色会突然变成一样的(因为它们此时具有相同的beta值)。原子选择中的原子指的是原始分子中的原子。当你通过选择改变一些原子的属性(如beta值),这些变化会反映在所有其它包括这些原子的选择中。
6 现在,输入set sel [atomselect top "hydrophobic"]。 这新建了一个包括所有疏水残基的选择。
7 让我们把所有的疏水原子的beta值设为1来标记它们。 你现在应该明白如何操作了:$sel setbbeta 1。如果OpenGL Display中的颜色没有更新,单击Graphical Representations底部的Apply按钮。
原子属性的例子。你可以通过应用原子选择,包括不同部分、残基、原子名、位置(x,y和z),电荷、质量、体积、半径等等来获得或者设置许多原子的属性。
8 现在要通过改变原子的物理性质来进一步说明疏水基团的分布。 输入$crystal set radius1.0,以使所有的原子变得小一点,更易观察。输入$sel set radius 1.5,让疏水基团大一点。半径的大小范围会影响图形的显示方式(如VDW, CPK)。现在已经新建了一个图像,可以清楚地区别蛋白质的疏水部分和亲水部分。如果你完全按照教程的要求来做,你得到的蛋白质图像就会如图12所示。
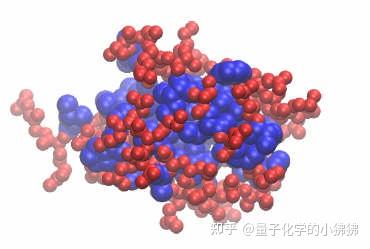
疏水残基。就像你在渲染泛素分子的过程中可能注意到的那样,疏水残基绝大部分都处于蛋白质的核心区。这在小的可溶性蛋白质中是很典型的。当蛋白质折叠的时候,疏水残基就有停留在界面的趋势。这样疏水基团汇集在一起,扮演着结构上的角色。这可以帮助蛋白质正确折叠,并提高稳定性。
原子选择不止在设置原子数据方面有用,而且可以用来获取信息。如果你想得到疏水基团,你要做的只是新建一个疏水选择,这里应该用get,而不是set。
9 在你选择的疏水原子上应用get
$sel get resname
但这里有个问题。每个残基都包含了很多原子,这样导致的结果是有很多重复输入。你能想到一个办法来克服这个问题吗?我们知道,每个氨基酸都有同样的骨架原子。如果你在每个残基中只选择一个这样的原子,每个残基就只会在你的选择中出现一次。
10 让我们试试这种解决办法。每一个原子有且只有一个α-碳(命名CA = alpha):
set sel [atomselect top "hydrophobic and alpha"]
$sel get resname
哈,成功了!
11 你也可以同时得到多种属性。试试下面的命令:
$sel get resid
$sel get {resname resid}
$sel get {x y z}
12 一旦你完成了一个选择,可以很方便地用下面的命令来删除它们以节省内存开支:
$sel delete

